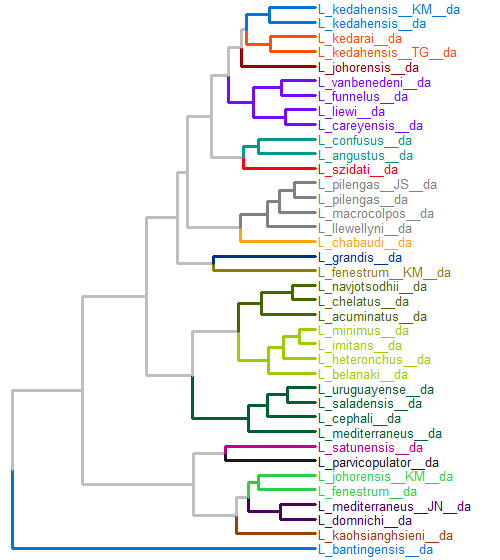
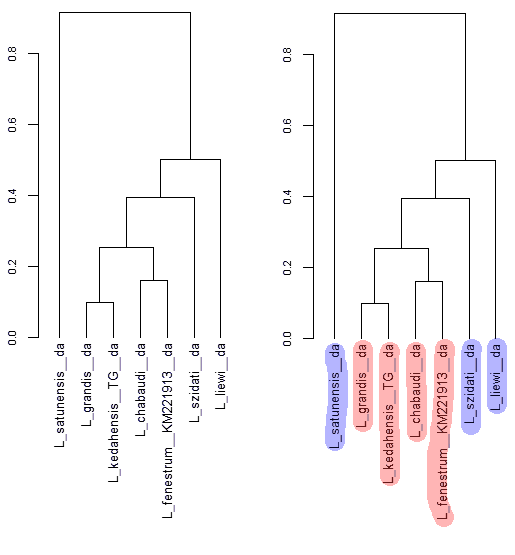
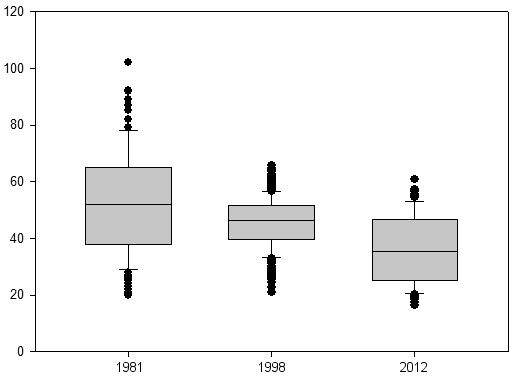
Дендрограмма — это результат объединения данных при помощи иерархического кластерного анализа. Обычно на листьях дендрограммы написаны имена переменных — названия объектов. Рассказываю, как вместо названий показать изображения.
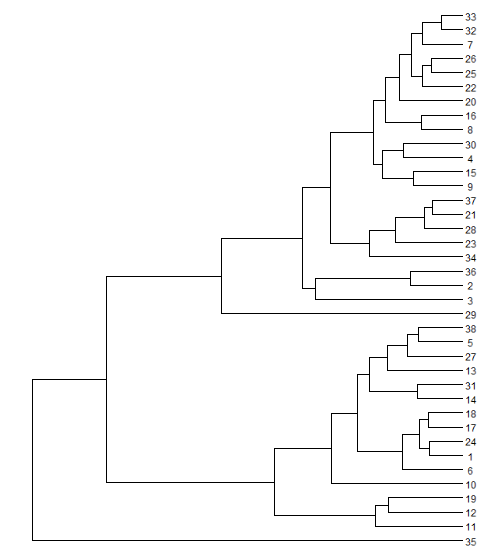
Эта красочная дендрограмма содержит 38 листьев и разделена на 19 классов. На листьях дендрограммы написаны названия видов плоских паразитических червей из класса моногеней. Собираюсь показать вместо них изображения прикрепительных органов этих паразитов
Для работы понадобятся
Знание языка Эр.
Установленный пакет ggtree.
Создаю изображения объектов
Каждому листу дендрограммы сопоставлю маленький рисунок объекта. Для этого буду использовать два набора изображений: один — для тестирования, другой — для финальной отрисовки.
Тестовые изображения
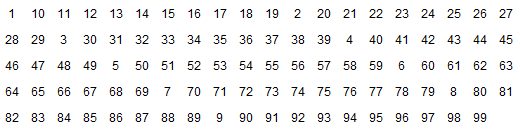
Тестовые изображения содержат номера, идущие по порядку. Они нужны для того, чтобы было понятно, куда именно разработанный код помещает картинки.
Вот код для генерации 99-ти пнг-файлов размером 20×20 пикселей. Имя файла с изображением совпадает с нарисованым номером.
imgs_path = "img_indexed/"
for (i in 1:99) {
# Создаю новый пнг-файл img_{i}.png с нулевыми отступами
png(file = paste0(imgs_path, "img_", i, ".png"), width = 20, height = 20)
par(mar = c(0, 0, 0, 0))
# Созданию рисунок числа
plot(0, 0, type = "n", xlim = c(0, 1), ylim = c(0, 1), xlab = "", ylab = "", axes = FALSE)
text(0.5, 0.5, i, cex = 1)
# Сохраняю пнг-файл
dev.off()
}
Вот сами изображения.
Вод код для их загрузки.
imgs_indexed = c()
imgs_path = "imgs_indexed/"
for(i in 1:75) {
imgs_indexed = c(imgs_indexed, paste0(imgs_path, "img_", i, ".png"))
}
Изображения объектов
Изображение объекта — это уменьшенный рисунок прикрепительного крючка паразитического червя (моногенеи). Вот они все:
Название файла с крючком совпадает с наванием вида. Эти названия храню списком в текстовом файле labels.txt. Для загрузки изображений объектов использую следующий код.
imgs_path = "img_objects/"
# Загружаем имена меток
img_objects <- readLines("labels.txt")
# и добавляем расширение '.png'
img_objects <- paste0(imgs_path, img_objects, ".png")
Изображения готовы, перейдем к дереву.
Строю дерево с картинками, используя ggtree
В книге «Data integration, manipulation and visualization of phylogenetic trees» описано, как прикрутить картинки к дендрограмме с помощью ggtree.

К сожалению, приведнный в книге код у меня не заработал. Он подразумевает, что мы загружаем филодерево из файла. А я строю дерево при помощи иерархической кластеризации и преобразую ее в объект дендрограммы.
data — это матрица данных: в ее строках — объекты, в столбцах — признаки.
# Выполняю иерархический кластерный анализ
dist <- dist(data)
hc <- hclust(dist, method = "complete")
library(dendextend)
# Преобразую результат в дендрограмму
dend <- as.dendrogram(hc)
Из дендрограммы делаю филодерево.
library(ggtree)
phylo <- as.phylo(dend)
Рисую дерево.
ggtree(phylo)
И получаю пустой каркас.
Строю дендрограмму с текстовыми листьями
Добавлю к листьям каркаса текстовые подписи при помощи функции geom_tiplab(∙). Чтобы подписи поместились на канве, оставлю пустое место справа при помощи xlim(∙).
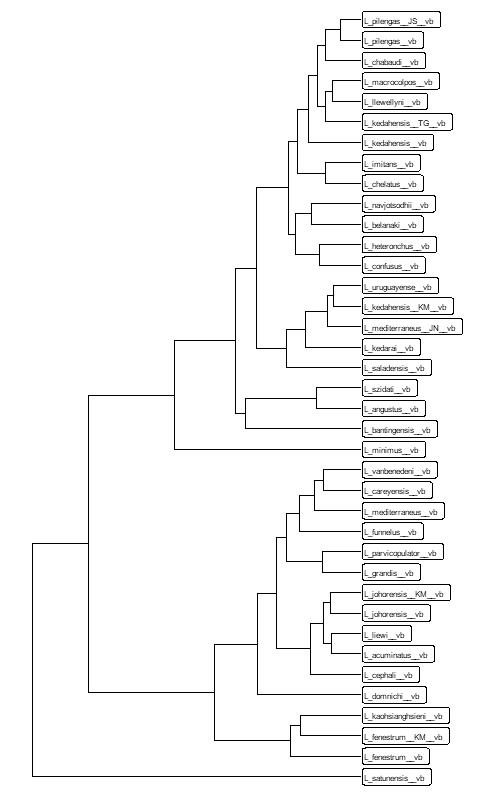
ggtree(phylo) + xlim(0, 0.7) + geom_tiplab(geom="label", size=2.2)
R: ggtree(∙) + xlim(∙) + geom_tiplab(label)
Получилось так себе: текст мелкий и бесит рамка вокруг него, но как от нее избавиться — не понял.
Метки мелкие и иногда перекрывают друг на друга. От перекрытия меток избавит пакет ggrepel. Но в результате получается каша.
Рисую картинки на дендрограмме
У функции geom_tiplab(∙) есть параметр geom, задающий тип метки:
one of ’text’, ’label’, ’shadowtext’, ’image’ and ’phylopic’.
При помощи опции ’image’, можно на месте листьев нарисовать картинки.
На нашей дендрограмме 38 листьев. Значит передадим ей массив из 38 тестовых изображений: imgs_indexed[1:38].
ggtree(phylo) + geom_tiplab(geom="image", aes(image=imgs_indexed[1:38]), size=.03)
В результате выполнения кода получил ошибку:
Error in `label_geom()`:
! Problem while computing aesthetics.
i Error occurred in the 3rd layer.
Caused by error in `check_aesthetics()`:
! Aesthetics must be either length 1 or the same as the data (75)
x Fix the following mappings: `image`
После длительного анализа определил, что массив изображений должен состоять из 75 элементов, что написано в сообщении об ошибке. (75 = 38 листьев * 2 − 1.) Но код все равно использует только 38 первых изображений. (Это хорошо.)
ggtree(phylo) + geom_tiplab(geom="image", aes(image=imgs_indexed[1:75]), size=.03)
Вуаля.
R: ggtree(∙) + xlim(∙) + geom_tiplab(image)
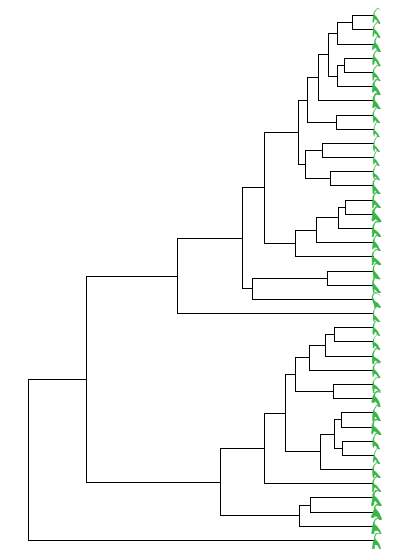
Подставлю сюда изображения объектов.
ggtree(phylo) + geom_tiplab(geom="image", aes(image=img_objects[1:75]), size=.03)
Цель достигнута.
Финальный код
library(ggplot2)
library(ggtree)
# Формируем массив с названиями файлов с рисунками объектов.
imgs_path = "img_objects/"
img_objects <- readLines("labels.txt")
img_objects <- paste0(imgs_path, img_objects, ".png")
# Выполняю иерархический кластерный анализ
dist <- dist(data)
hc <- hclust(dist, method = "complete")
# Преобразую результат в филодерево
phylo <- as.phylo(dend)
# Рисую дерево с картинками
ggtree(phylo) + geom_tiplab(geom="image", aes(image=img_objects[1:75]), size=.03)
Итог
Результат не нравится.
Дендрограмма вверху заметки — красивая, а эта — средненькая. Поэтому в следующий раз покажу, как сделать красивую дендрограмму с картинками при помощи пакета dendextend.
Если после прочтения этой прекрасной заметки вам вдруг непреодолимо захотелось меня поблагодарить, переведите мне 200 руб. на круасан с чаем. А я пока напишу что-то новенькое.













-Mnogomerny-dispersionny-analiz.png)
-Blok-shema-mnogomernogo-disp-i-diskr-analizov.png)
-Primer-formul---2.png)
-Primer-formul.png)
---Dispers-analiza-na-EVM.png)
---primer-2.png)
---primer.png)
-Disp-analiz.gif)
---primer.png)
---primer-2.png)
---Oblozhka.png)
---Uboy-bychkov.png)
---Schelknite-OK.png)
