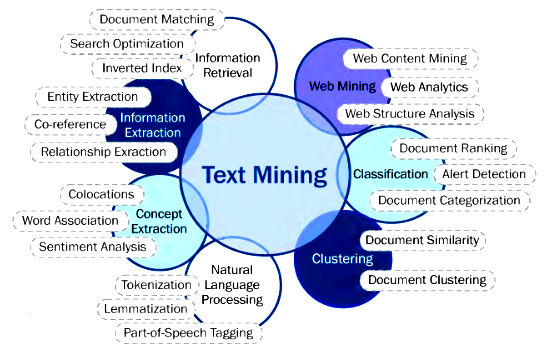
Для работы мне потребовался список всех известных к настоящему времени родов диатомовых. Вот результаты попыток его получить.
Статьи
Рода диатомовых перечислены в некоторых публикациях. Например, в каталоге родов диатомовых 1999 года
Fourtanier E. & Kociolek J.P. (1999) Catalogue of diatom genera.
Из которого вырос каталог названий диатомовых 2009 года, тех же авторов.
Fourtanier E. & Kociolek J.P. (2009) Catalogue of diatom names.
Но мне нужен электронный список — текстовый файл или отформатированная таблица. Извлечь список из пдф статей — сложная задача. Поэтому этот вариант мне не подходит.
Базы данных
Очевидно, что в наш век, когда нейросети потихоньку покоряют человечество, найти в интернете базу данных с полным списком родов определенного таксона не составляет никакого труда. И да, такие базы есть.
Почти все названия диатомовых хранят:
При этом Диатомбейз не является независимой базой — она извлекает данные из базы Вормса и копирует его интерфейс. То есть ее можно спокойно вычеркнуть, оставив Альгабейз и Вормс.
Ни одна из оставшихся двух баз, ни Альгабейз, ни Вормс, не позволяет просто взять и получить список таксонов определенного ранга.
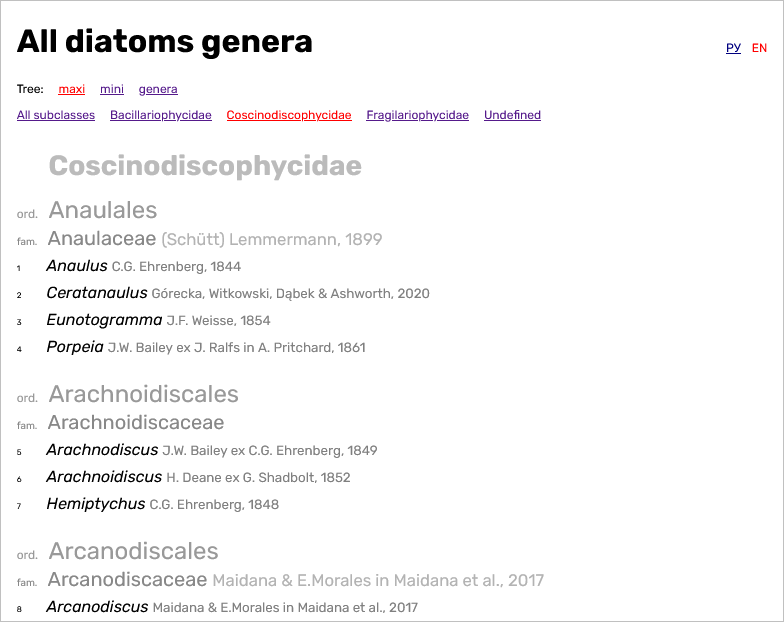
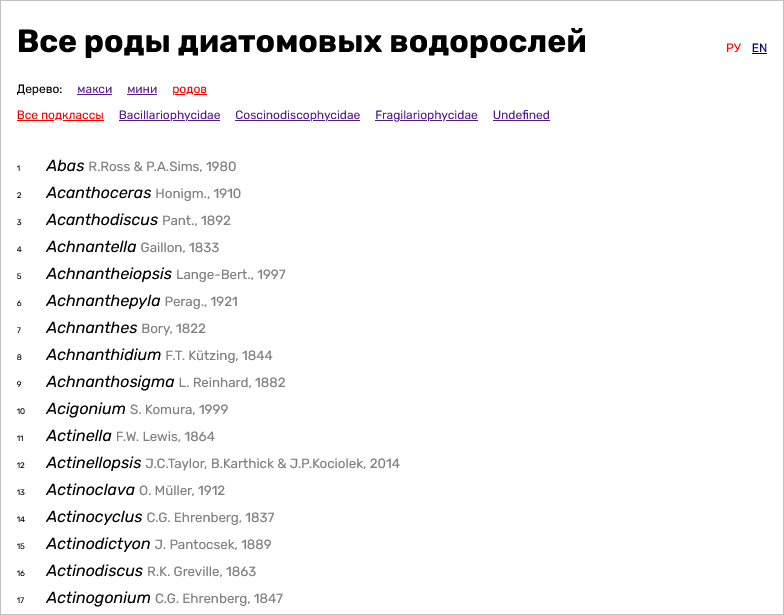
Для этого необходимо пробежать по дереву: от верха до нужного ранга, раскрывая по дороге все боковые ветви: над- и под-отделы и отделы, над- и под-классы и классы, надсемейства и семейства. Все без исключения.
При этом страничка с перечнем родов определенного высшего таксона представляет собой сложноструктурированный текст, из которого тоже не очень-то просто вычленить список родов.
То есть вариант использования баз, в котором нужно ручками пролистать ветви таксономического дерева и повыковыривать названия родов тоже мне не подходит.
АПИ
Альгабейз не предоставляет никакой иной официальной возможности доступа к базе, кроме как через интерфейс веб-страницы. Майкл Гури зорко охраняет собранные таксономические данные.
К счастью Вормс — это открытая система. Он предоставляет АПИ для извлечения данных.
Но именно тот запрос, который должен вернуть названия всех таксонов заданного ранга, относящихся к конкретному родителю, в данный момент работает неправильно.
/AphiaRecordsByTaxonRankID/{ID}
Get the AphiaRecords for a given taxonRankID (max 50)
Сейчас он возвращает только первые 50 названий (родов). Остальные остаются недоступными.








---title.png)
---title.png)